| Ubicación: | Laboratorio 14 UBIMED |
| Teléfono: | 55 562 31333 ext. 39785 |
| Responsable del Laboratorio: | Dr. Luis Enrique Arias Romero Profesor Titular “B” de Tiempo Completo larias@unam.mx cancercellsignaling.unam@gmail.com |
| Académicos incorporados al laboratorio: | Dr. Ricardo Hernández García mazing11@hotmail.com M. en C. Héctor Iván Saldivar Cerón ivansaldi@msn.com Biol. Tania Reyes Mendoza taniareyes516@hotmail.com José Manuel Hernández Cruz manuelhcr3@gmail.com |
| Técnicos académicos incorporados al laboratorio: | M. en C. Olga Villamar Cruz Técnico Académico Titular “C” de Tiempo Completo vicovc@gmail.com |
| Líneas principales de los académicos incorporados: | Dr. Ricardo Hernández García: Regulación de la actividad del RhoGEF Vav1 por la proteína fosfatasa de tirosinas PTP1B. M. en C. Olga Villamar Cruz: Estudio de la ruta de señalización PTP1B-CDK3 en células de glioblastoma. M. en C. Héctor Iván Saldivar Cerón: Identificación de nuevos sustratos de la proteína cinasa PAK1 en cáncer de mama. Biol. Tania Reyes Mendoza: Identificación de nuevos sustratos de la proteína cinasa PAK1 en cáncer de mama. |

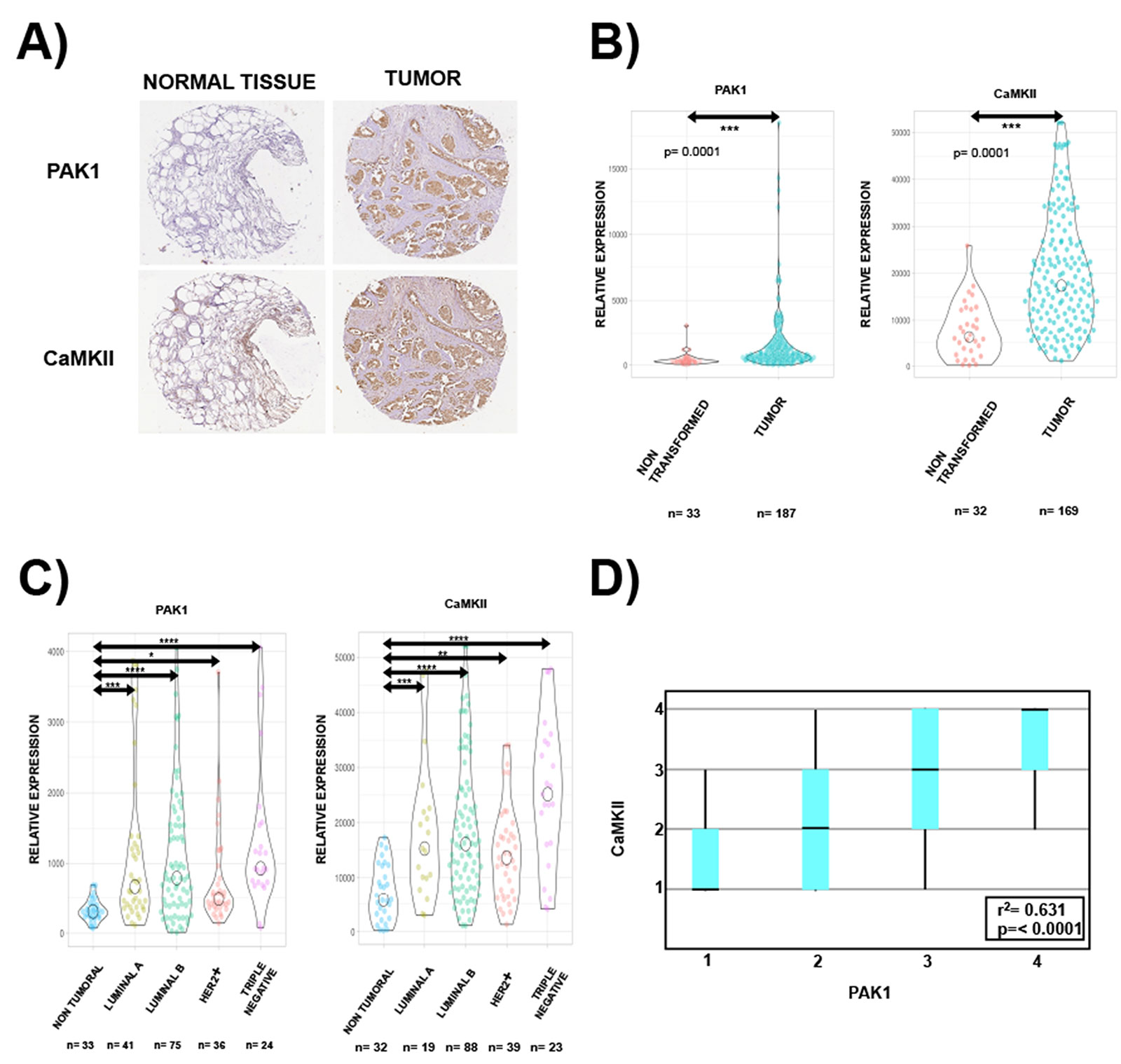
La investigación de nuestro grupo está enfocada en el estudio de las rutas de señalización mediadas por el receptor HER2 en cáncer de mama. En particular, estamos interesados en identificar elementos río abajo de HER2 que pudiesen ser empleados como nuevos blancos de intervención terapéutica.
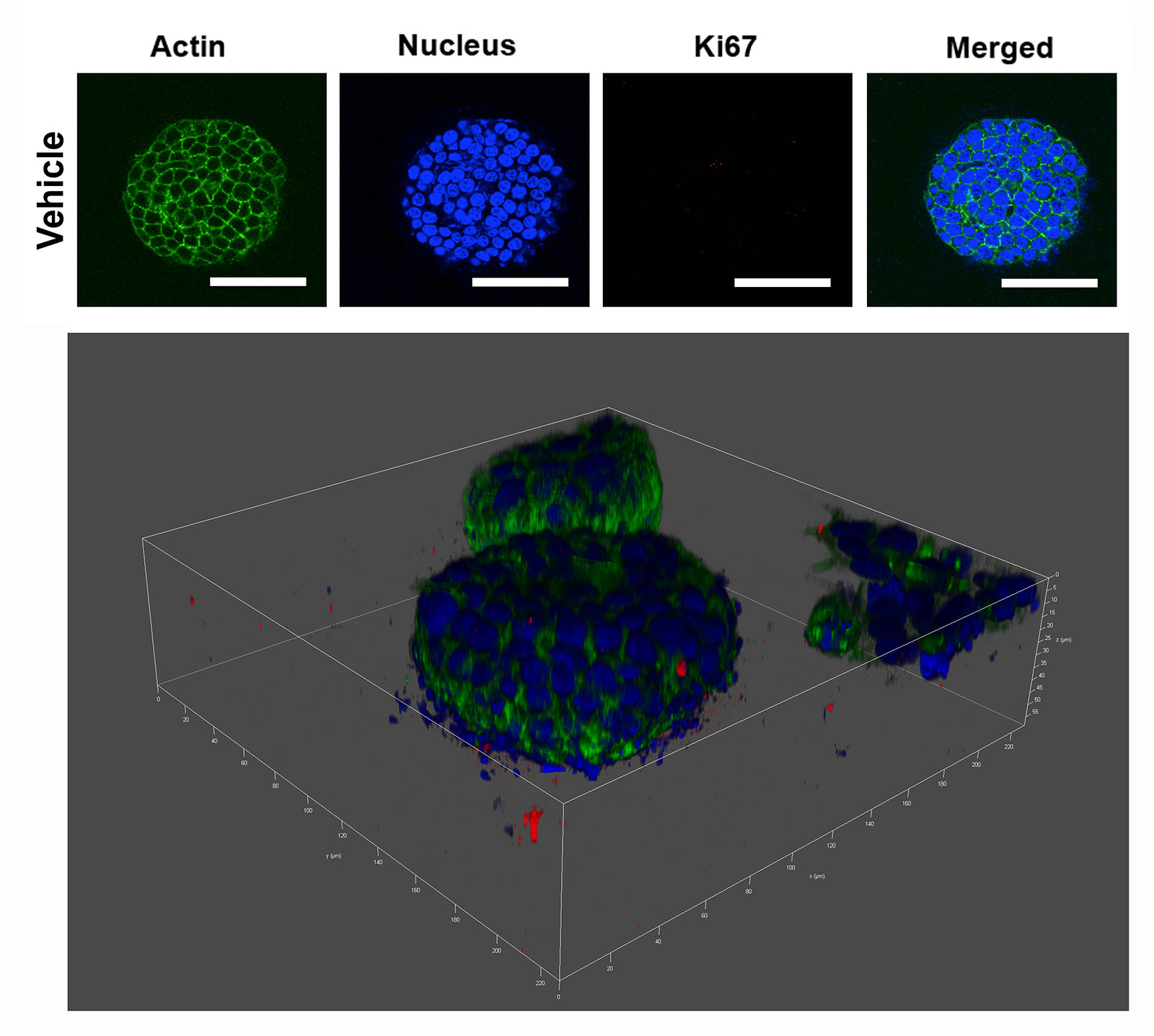
Uno de estos elementos es el eje de señalización formado por la GTPasa pequeña Rac1, y uno sus efectores, la proteína cinasa PAK1. Para estudiar de manera detallada los mecanismos moleculares por los cuales estas moléculas promueven la transformación celular, utilizamos un sistema de cultivo en 3 dimensiones (3D) que recapitula el desarrollo de los ductos de la glándula mamaria.
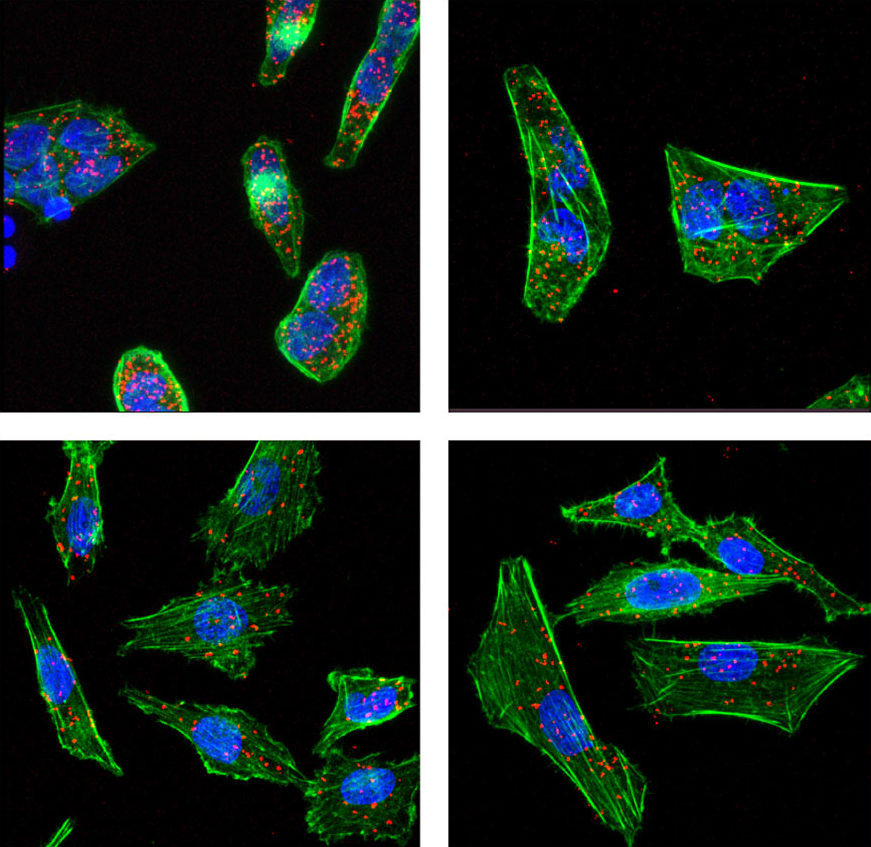
Adicionalmente, el uso de técnicas de fosfoproteómica, nos ha permitido identificar diversas proteínas señalizadoras que pudieran estar reguladas directa o indirectamente por PAK1, y que tienen el potencial de ser empleadas como nuevos blancos de intervención terapéutica.
Estos cultivos ofrecen la ventaja de poder ser analizados bioquímica y celularmente por técnicas convencionales como inmunofluorescencia y western blot. Además de que cuando se sobre expresa algún oncogén, las estructuras 3D muestran características similares a las del carcinoma ductal in situ, como son perdida de la polaridad y de los contactos célula-célula, inhibición de la apoptosis e incremento de la proliferación celular.
Actualmente, nos encontramos estudiando algunos de los potenciales nuevos sustratos de PAK1 empleando técnicas bioinformáticas, docking, técnicas bioquímicas y de biología celular, así como el impacto de la inhibición individual o combinada de PAK1 y sus potenciales sustratos en diversos procesos celulares, así como en el desarrollo y progresión tumoral en modelos animales.

1) Arias-Romero LE, Saha S, Villamar-Cruz O, Yip SC, Ethier SP, Zhang ZY, Chernoff J. (2009). Activation of Src by Protein Tyrosine Phosphatase 1B Is Required for ErbB2 Transformation of Human Breast Epithelial Cells. Cancer Res, 69(11): 4582-88. doi: 10.1158/0008-5472.CAN-08-4001
2) Arias-Romero LE, Villamar-Cruz O, Huang M, Chernoff J. (2013). p21-activated kinase-1 links ErbB2 to β-catenin in transformation of breast epithelial cells. Cancer Res, 73(12):3671-82. doi: 10.1158/0008-5472.CAN-12-4453
3) Rawat SJ, Araiza-Olivera D, Arias-Romero LE, Villamar-Cruz O, Prudnikova TY, Creasy CL, Chernoff J. (2016). H-ras inhibits the hippo pathway by promoting Mst1/Mst2 heterodimerization. Current Biol, 26(12): 1553-63. doi: 10.1016/j.cub.2016.04.027
4) Villamar-Cruz O, Prudnikova TY, Araiza-Olivera D, Perez-Plasencia C, Johnson N, Bernhardy AJ, Slifker M, Renner C, Chernoff J, Arias-Romero LE. (2016). Reduced PAK1 activity sensitizes FA/BRCA-proficient breast cancer cells to PARP inhibition. Oncotarget, 7(47):76590-603. doi: 10.18632/oncotarget.12576
5) Saldívar-Cerón HI, Villamar-Cruz O, Pérez-Plasencia C, Arias-Romero LE. (2018). p21 Activated kinase 1: Nuclear activity and its role during DNA damage repair. DNA Repair, 65:42-46. doi: 10.1016/j.dnarep.2018.03.004

| Perfil de ingreso de potenciales tesistas: | Tesistas de las carreras de Biología, Q.F.B y afines con interés en biología celular, bioquímica y biología molecular del cáncer. |
| Datos de Servicio Social: | Programa: Señalización celular en cáncer. Clave de Registro: 2020-12/63-60 Objetivo: Identificación de moléculas señalizadoras con potencial terapéutico. Actividades: Cultivo de bacterias, purificación y análisis de ácidos nucleicos y proteínas, cultivo celular, búsqueda de información en bases de datos, entrega de reportes. |